编辑 | 萝卜皮
准确识别蛋白质-ATP 结合位姿对于基础结构生物学和药物发现都具有重要意义。尽管已经设计了许多对接方法,但大多数都需要用户定义的结合位点,很难达到高质量的蛋白质-ATP对接结果。开发一种没有用户定义的结合位点的蛋白质-ATP 特异性盲对接方法至关重要。
在这里,浙江工业大学的研究人员介绍了 ATPdock,这是一种基于模板的方法,用于将 ATP 与蛋白质对接。
对于每个查询蛋白,如果没有给出口袋位点,ATPdock 首先使用 ATPbind(一种 ATP 结合位点预测器)识别其最有潜力的口袋;然后,使用口袋结构对齐工具 APoc 从口袋-配体结构数据库中搜索与给定或识别口袋最相似的模板口袋;第三,使用配体结构对齐工具LS-align生成ATP的粗对接位姿(rdATP),将初始ATP位姿与模板口袋对应的模板配体对齐;最后,在 AutoDock Vina 能量函数的指导下,利用 Metropolis Monte Carlo 模拟对 rdATP 进行微调。基准测试表明,ATPdock 在对接精度方面明显优于其他最先进的方法。
该研究以「ATPdock: a template-based method for ATP-specific protein–ligand docking」为题,于 2022 年 1 月 15 日刊载在《Bioinformatics》。

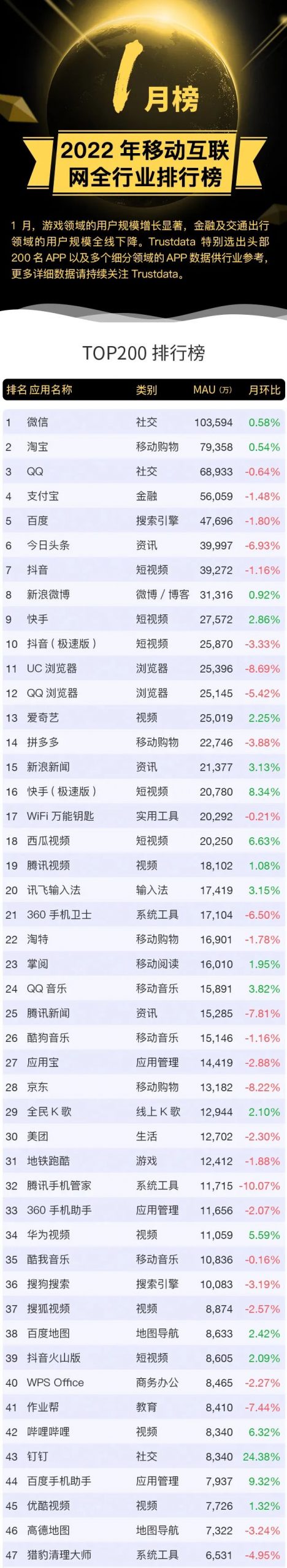
ATPdock的性能评估
ATPdock 使用两个最先进的蛋白质-配体对接程序(AutoDock Vina 和 DOCK6)在 88 种蛋白质-ATP 复合物上进行了测试。在 88 个复合物中有 94 个天然和 88 个 ATPbind 预测的口袋 ATP 结合位姿。
使用 APoc 和 LS-align 算法,对于每个查询口袋,ATPdock 可以从 PLDB 数据库中搜索具有最高相似性的模板口袋-配体结构。结果显示了在提供原生和 ATPbind 预测的口袋信息的条件下相应的相似性值。
鉴于原生 ABP 信息,ATPdock 在 94 个口袋 ATP 结合姿势上的平均 RMSD 值可以达到 3.41 Å,分别比 AutoDock Vina 和 DOCK6 低 53.9% 和 45.3%。
鉴于 ATPbind 预测的 ABP 的信息,ATPdock 的平均 RMSD 值比两个控制程序,即 AutoDock Vina 和 DOCK6 的平均 RMSD 值低 39.8% 和 40.2%。
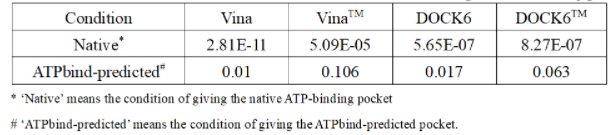
此外,通过重新访问图 1A 和 B,很容易看出从 PLDB 数据库中搜索的口袋配体模板可用于帮助提高 AutoDock Vina 和 DOCK6 的性能。
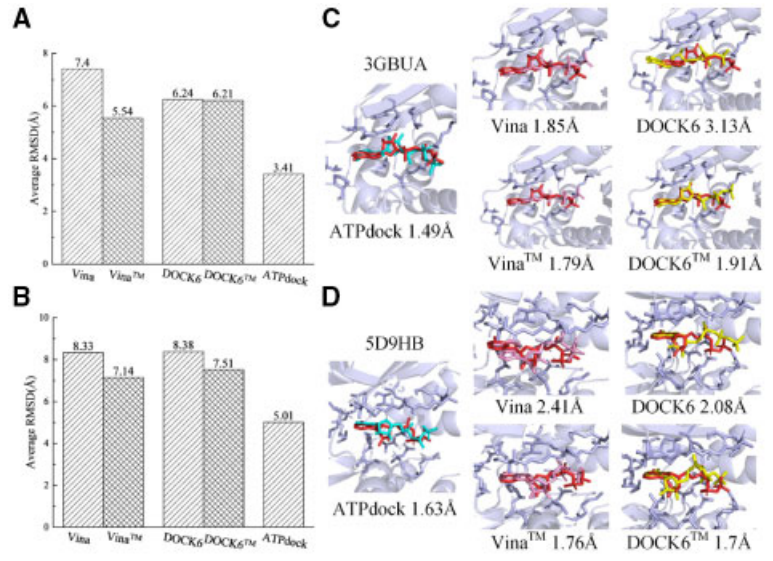 图1:ATPdock的性能评估。(来源:论文)
图1:ATPdock的性能评估。(来源:论文)表 1 显示了 Student t-test 中 ATPdock 和 Vina、VinaTM、DOCK6 和 DOCK6TM 在两种不同给定信息上的 RMSD 差异的 P 值。从表 1 中不难看出,在给出原生口袋信息时,ATPdock 与其他对接方法之间的 P 值均 <10^4。这里,上标 TM 表示 rdATP 在相应的对接方法中用作初始状态。
表1:在两种不同给定信息上的 RMSD 差异 P 值。(来源:论文)
此外,图 1C 提供了一个说明性示例,将 ATP 对接到查询蛋白的天然口袋(PDB ID:3GBUA),其中 ATPdock 的对接结果在所有对接结果中具有最低的 RMSD(1.49 Å)。图 1D 显示了将 ATP 对接到查询蛋白的 ATPbind 预测口袋中的另一个示例(PDB ID:5D9HB),其中 ATPdock 的对接结果在所有对接结果中具有最低的 RMSD(1.63 Å)。图 2 显示了在给出天然和 ATPbind 预测口袋的条件下 RMSD 的分布。
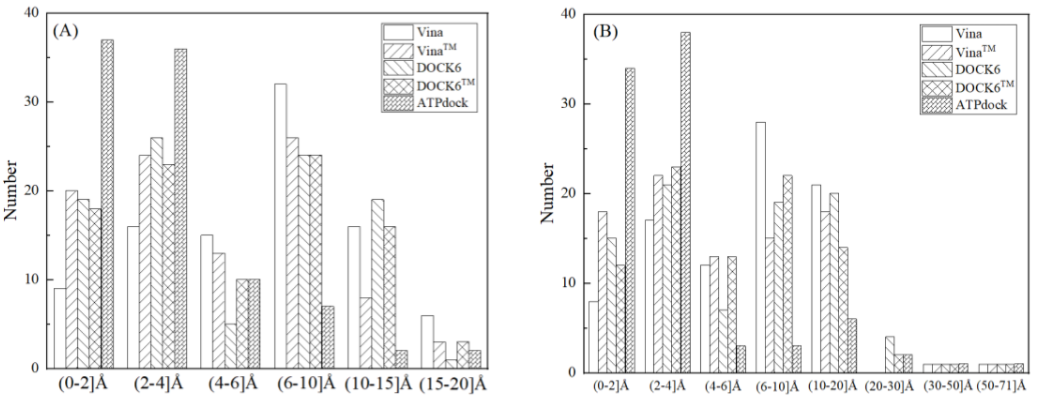
图 2:ATPdock、Vina、DOCK6、VinaTM 和 DOCK6TM 在原生和 ATPbind 预测口袋条件下对接结果的 RMSD 分布。(来源:论文)
实际上,搜索到的口袋配体模板的质量越高,ATPdock 的性能就越好。除了使用的口袋配体模板外,ATPdock 的高对接精度可部分归因于 Metropolis Monte Carlo 模拟。图 3 显示了一个案例研究,其中使用 Metropolis Monte Carlo 的模拟导致与没有 Metropolis Monte Carlo 程序的完全不同的对接姿势;前者的 RMSD 比后者低 21.6%。
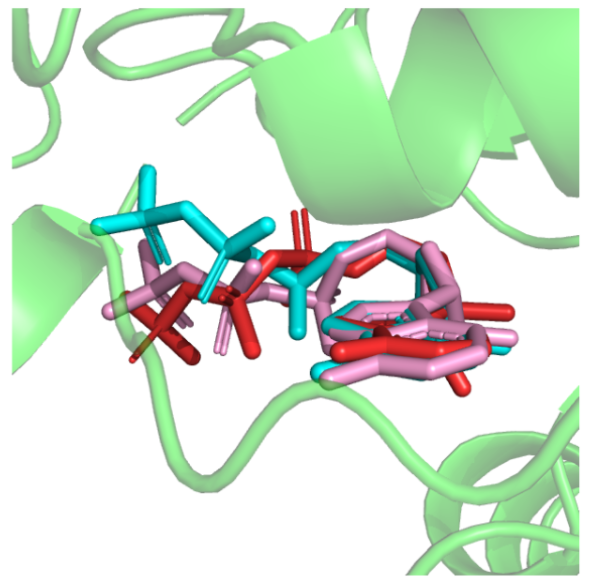 图 3:ATPdock在5DB4A上的对接结果。(来源:论文)
图 3:ATPdock在5DB4A上的对接结果。(来源:论文)ATPdock 的网络服务器
唯一的强制性输入是 PDB 格式的查询蛋白质结构信息。ATPdock 的网络服务器还允许用户可选地输入一个或多个 ABP 信息以生成一个或多个相应的 ATP 绑定姿势。如果没有输入 ABP,使用 ATPbind 生成一个或多个 ABP 需要额外的几个小时。
对于每个 ATPdock 生成的 ATP 绑定姿势,由 NGL Viewer 实现的四个交互式小程序,用于可视化查询和搜索模板袋的叠加结构,初始 ATP 和搜索的模板配体的叠加结构,查询口袋和初始 ATP 的粗对接结构,通过将它们与搜索的口袋-配体模板对齐获得,以及最终对接结构。
ATPdock 网络服务器可以轻松扩展以将 ATP 样(甚至通用)配体停靠到受体蛋白中。未来,研究人员还应该使用其他搜索方案来快速找到 ATP 或其他配体类型的最佳对接姿势。
ATPdock 独立程序:https://github.com/jun-csbio/ATPdock
ATPdock 网络服务器和数据集:https://jun-csbio.github.io/atpdock/
论文链接:https://academic.oup.com/bioinformatics/article-abstract/38/2/556/6373371?redirectedFrom=fulltext&login=false