▎药明康德内容团队编辑
本周,顶尖学术期刊《自然》在线发表了一篇重量级研究。在华盛顿大学(University of Washington)蛋白设计研究所David Baker教授的主导下,曹龙兴博士与同事们展示了一种极具创新的蛋白设计策略:可以针对几乎任何感兴趣的靶点蛋白,利用计算机软件设计出与之紧密结合的蛋白分子作为候选药物,其应用可广泛覆盖从癌症到新冠感染的诸多疾病。
David Baker教授是人工设计蛋白领域的领军人物,其团队开发的一款名为Rosetta的程序,能根据蛋白质的氨基酸序列有效预测蛋白质的结构,在此基础上可以从头设计各种类型的全新蛋白质。
但有一类蛋白的构建一直是巨大的挑战,那就是在癌症等重大疾病的治疗方法中常用到的“结合蛋白”:它们与疾病相关的靶点蛋白相互作用,改变靶点蛋白的功能。人们熟悉的抗体药物就属于这类蛋白,与癌细胞特有的表面蛋白结合,或是与入侵微生物结合,从而发挥抗癌或抗感染的作用。



▲研究论文的通讯作者David Baker教授和共同第一作者曹龙兴博士和Brian Coventry博士(图片来源:参考资料[2])
不过,抗体作为大分子蛋白质,制造难度和稳定性都给应用带来一定局限。为了解决这个问题,一种思路是制造小分子的结合蛋白,这些“迷你版抗体”同样可以结合在靶标蛋白的特定位置,但由于尺寸更小,通常更稳定、更便于生产,只是设计的难度非常高。
研究人员打了一个比方:假如把靶标蛋白质想象成一面攀岩墙,那么其氨基酸序列经过折叠形成的褶皱和凸起相当于攀岩支点,为攀爬提供双手的握点和脚的登点。大分子的抗体可以同时抓住好多支点,稳稳爬到顶点。而小分子的“攀岩者”可选择的支点就少多了,必须找到最佳的握点和登点,规划出登顶路线。
曹龙兴博士与同事们借助Rosetta设计的新方法则是这样的:找出攀岩墙上大大小小每一个握点和登点,让成千上万的攀岩者尝试各种可能的路线,然后新的攀岩者在最有希望的路线上得出最佳路径。“只需要几个小时的计算就可以尝试各种可能的路线。”曹博士说。
2020年爆发新冠疫情后,在Baker教授的主导下,曹龙兴博士与同事们利用这种策略设计了可以结合新冠病毒刺突蛋白的小型蛋白。他们在计算机上设计了超过200万个候选的结合蛋白,最终找到抗病毒活性最强的一种结合蛋白,可以像单克隆抗体一样阻止新冠病毒感染。
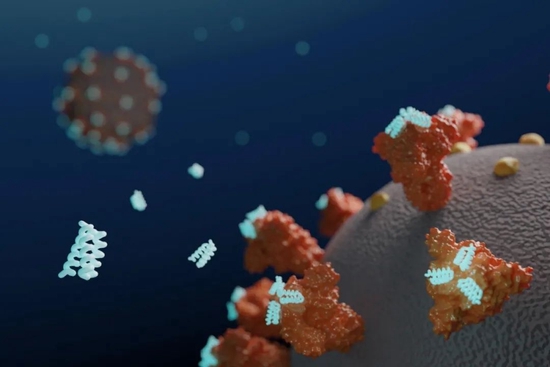
▲学术经纬相关阅读:“科学突破奖”新得主再迎突破,人造蛋白可抗新冠病毒(图片来源:参考资料[2];Crdit:UW Medicine Institute for Protein Design)
小试牛刀之后,研究团队在此次研究中进一步扩展了范围,给12种不同的靶标蛋白设计小分子结合蛋白。这些靶标包括参与癌症发生发展的生长因子、胰岛素受体、流感病毒等病原体的表面蛋白等,生物学功能多样,形状各异,具有不同表面化学特性。
研究人员针对每种靶标蛋白质的多个结合位点设计了数万个虚拟的小型结合蛋白进行筛选,并通过进一步修饰增加结合蛋白与靶点的亲和力,利用酵母将人工设计的蛋白制造出来。这些小型结合蛋白不超过65个氨基酸,尺寸只有一般单克隆抗体的1/20。而经过测试,最终他们为所有12个靶标蛋白都设计出了亲和力与抗体可媲美的小型结合蛋白。
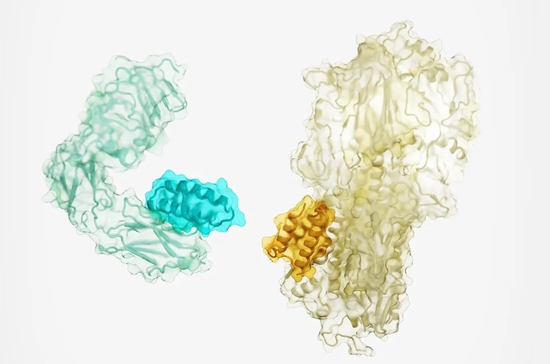
▲与胰岛素受体(左)和流感病毒血凝素(右)结合的小型结合蛋白(图片来源:参考资料[3];Credit:Ian C Haydon / UW INSTITUTE FOR PROTEIN DESIGN)
“我们可以针对自然界任意蛋白质设计全新结合蛋白。”曹龙兴博士说。这一结果不仅验证了设计方法广泛的适用性,也显示出这些小型结合蛋白作为药物的潜力。
研究论文发表后,《科学》杂志也专门报道了这一进展,多位没有参与该研究的专家给予了很高的评价,加州大学旧金山分校的生物工程学家Tanja Kortemme教授认为这一方法“开辟了许多新的可能”。Baker教授则指出,他们还将继续改进计算方法,以便人工设计的小型结合蛋白更好地满足疾病治疗的需求。
参考资料:
[1] Longxing Cao et al。 (2022) Design of protein binding proteins from target structure alone。 Nature https://doi.org/10.1038/s41586-022-04654-9
[2] Design of protein binding proteins from target structure alone。 Retrieved Mar。 25, 2022 from https://endpts.com/a-major-step-forward-scientists-unveil-new-approach-for-designing-protein-drugs-against-any-target/
[3] Software-designed miniproteins could create new class of drugs。 Retrieved Mar。 25, 2022 from https://www.science.org/content/article/software-designed-miniproteins-could-create-new-class-drugs
[4] Antiviral proteins block coronavirus infection in the lab。 Retrieved Sep。 11, 2020, from https://www.ipd.uw.edu/category/coronavirus/